Por: Nayara Braga Emidio
Por que estudar proteínas de Schistosoma mansoni?
Entre os requisitos para que os agentes antiparasitários alcancem eficácia terapêutica estão: atuação em vias metabólicas exclusivas de parasitos e seletividade por proteínas específicas destes. Além dos quimioterápicos, o sistema imunológico também compõe um importante mecanismo de defesa contra os parasitos, sendo estimulado, principalmente, por proteínas.
Deste modo, a pesquisa de proteínas do verme, bem como seu grau de similaridade em relação as proteínas humanas e a maneira que tais moléculas atuam, constituem-se em um estudo de grande relevância.
Embora o genoma de S. mansoni já tenha sido sequenciado1, ainda tem-se pouco conhecimento sobre suas proteínas, e, principalmente, sobre a função destas no metabolismo do parasito.
Proteínas de Schistosoma mansoni
 As sequências e/ou anotações dos 12.871 genes de S. mansoni, assim como as de S. haematobium e S. japonicum, estão depositadas no banco de dados SchistoDB e são de livre acesso. Além disso, informações adicionais resultantes do processamento das sequencias e anotações do genoma, tais como domínios InterPro, termos de ontologia gênica, predição de peptídeo sinal, predição de domínios transmembranares, predição de ORFs (open reading frame), BLAST contra o banco de dados do NCBI, predição de genes ortólogos baseado no OrthoMCL e predição de sintenia genética, também podem ser encontradas no banco de dados. Assim, o SchistoDB, mais do que um depósito de sequências, é também um recurso de bioinformática útil no campo de pesquisa envolvendo esquistossomose2.
As sequências e/ou anotações dos 12.871 genes de S. mansoni, assim como as de S. haematobium e S. japonicum, estão depositadas no banco de dados SchistoDB e são de livre acesso. Além disso, informações adicionais resultantes do processamento das sequencias e anotações do genoma, tais como domínios InterPro, termos de ontologia gênica, predição de peptídeo sinal, predição de domínios transmembranares, predição de ORFs (open reading frame), BLAST contra o banco de dados do NCBI, predição de genes ortólogos baseado no OrthoMCL e predição de sintenia genética, também podem ser encontradas no banco de dados. Assim, o SchistoDB, mais do que um depósito de sequências, é também um recurso de bioinformática útil no campo de pesquisa envolvendo esquistossomose2.
Atualmente, 12.775 proteínas de S. mansoni estão depositadas no banco de dados UniProtKB, sendo que menos de 1% delas são revisadas, isto é, tem suas informações obtidas experimentalmente ou por análises computacionais criticamente avaliadas.
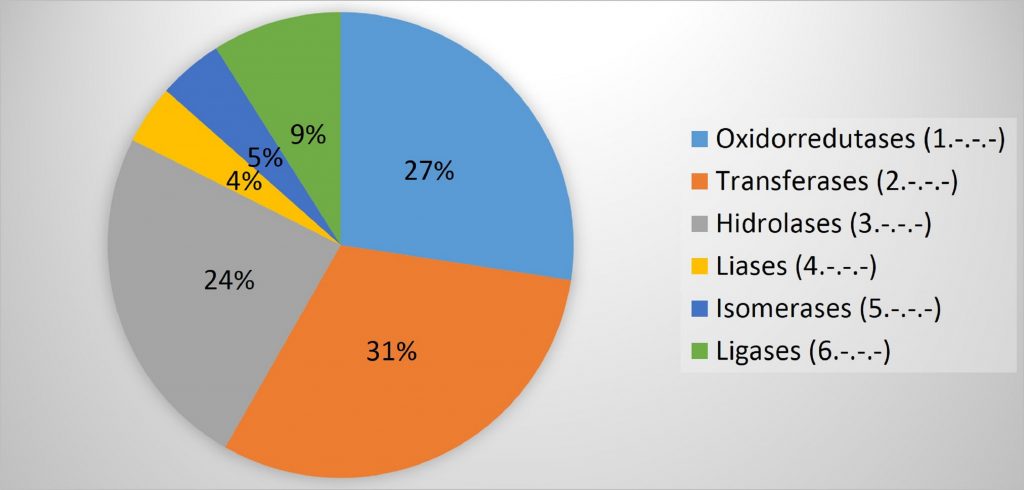
O gráfico abaixo mostra como as enzimas do verme disponíveis no UniProtKB estão divididas de acordo com a reação que catalisam3:
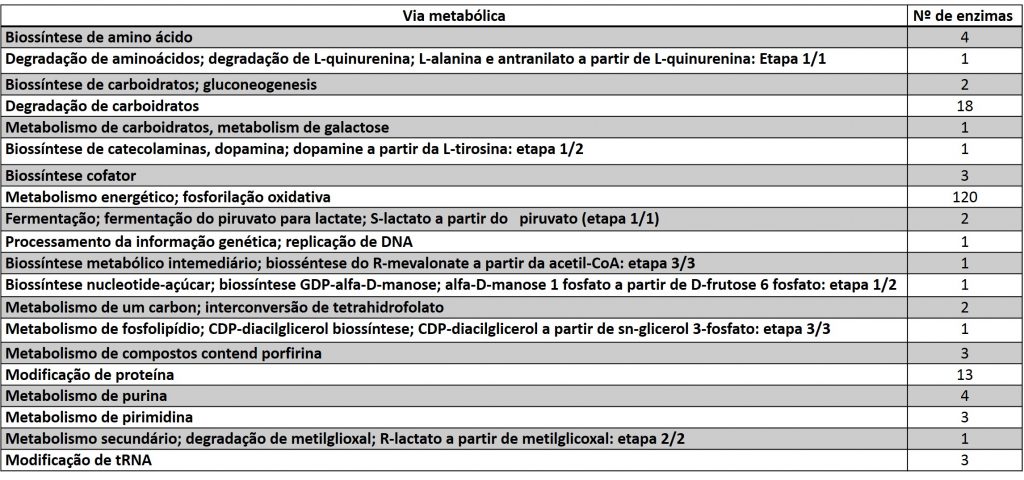
A tabela abaixo mostra como estas enzimas estão distribuídas de acordo com as vias metabólicas3:
Referências
1 A. V. Protasio, I. J. Tsai, A. Babbage, S. Nichol, M. Hunt, M. A. Aslett, N. De Silva, G. S. Velarde, T. J. Anderson, R. C. Clark, C. Davidson, G. P. Dillon, N. E. Holroyd, P. T. LoVerde, C. Lloyd, J. McQuillan, G. Oliveira, T. D. Otto, S. J. Parker-Manuel, M. A. Quail, R. A. Wilson, A. Zerlotini, D. W. Dunne, and M. Berriman, ‘A Systematically Improved High Quality Genome and Transcriptome of the Human Blood Fluke Schistosoma Mansoni’, PLoS Negl Trop Dis, 6 (2012), e1455. 
2 A. Zerlotini, E. R. Aguiar, F. Yu, H. Xu, Y. Li, N. D. Young, R. B. Gasser, A. V. Protasio, M. Berriman, D. S. Roos, J. C. Kissinger, and G. Oliveira, ‘Schistodb: An Updated Genome Resource for the Three Key Schistosomes of Humans’, Nucleic Acids Res, 41 (2013), D728-31.
3 Uniprot. Disponível em <http://www.uniprot.org/help/uniprotkb> Acessso: 08/04/2014